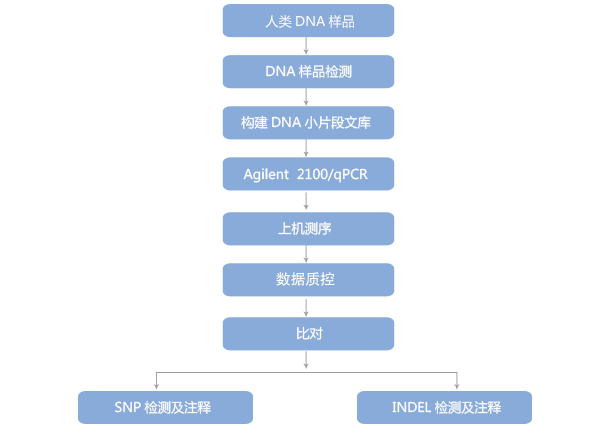
1、技术路线
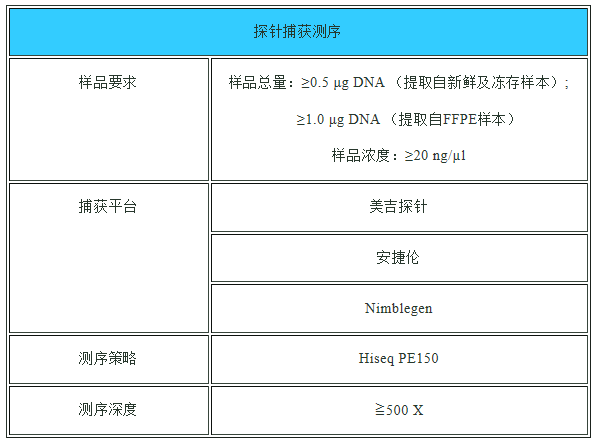
2、技术参数
3、案例解读
癌症个体化深度测序(CAPP-Seq)方法检测肺癌患者血浆ctDNA
研究背景:癌症个体化深度测序分析方法 (CAPP-Seq),对非小细胞肺癌 (NSCLC )的 ctDNA 检测结果灵敏度高,特异性强且经济可行。利用定制化的 NimbleGen SeqCap EZ Choice Library 作为"筛选器" (selector) 对样本进行靶向捕获后再进行深度测序。
研究结果:研究者从肿瘤基因突变数据库 (COSMIC) 等来源找出与 NSCLC 复发突变相关的外显子,再对来自肿瘤基因图谱 (The Cancer Genome Atlas) 数据库的 407 位 NSCLC 患者全基因组测序结果进行筛选,并应用迭代算法使筛选出的区域在充分覆盖每个样本的错义突变的同时尽可能的精简。罗氏NimbleGen根据以上区域定制了 NimbleGen SeqCap EZ Choice Library 靶向序列捕获产品作为本次 CAPP-Seq 的"筛选器",包含了 139 个相关基因的 521 个外显子和 13 个内含子,共约 125kb。 NimbleGen "筛选器"有效的把测序区段浓缩到整个基因组大小的 0.004%,使得后续超高深度测序得以实现。
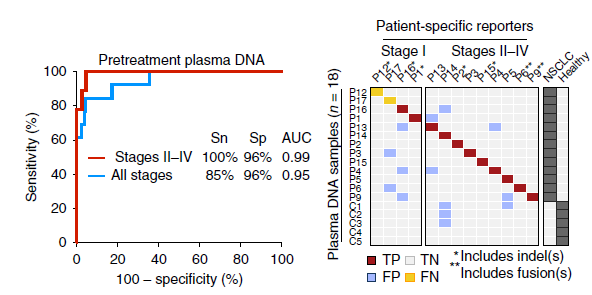
CAPP-seq 是一个相当好的检测肿瘤基因突变的方法
参考文献
Aaron M Newman,et al. An ultrasensitive method for quantitating circulating tumor DNA With broad patient coverage. Nature Medicine.2014 May;20(5):548-54.

2、技术参数

3、案例解读
癌症个体化深度测序(CAPP-Seq)方法检测肺癌患者血浆ctDNA
研究背景:癌症个体化深度测序分析方法 (CAPP-Seq),对非小细胞肺癌 (NSCLC )的 ctDNA 检测结果灵敏度高,特异性强且经济可行。利用定制化的 NimbleGen SeqCap EZ Choice Library 作为"筛选器" (selector) 对样本进行靶向捕获后再进行深度测序。
研究结果:研究者从肿瘤基因突变数据库 (COSMIC) 等来源找出与 NSCLC 复发突变相关的外显子,再对来自肿瘤基因图谱 (The Cancer Genome Atlas) 数据库的 407 位 NSCLC 患者全基因组测序结果进行筛选,并应用迭代算法使筛选出的区域在充分覆盖每个样本的错义突变的同时尽可能的精简。罗氏NimbleGen根据以上区域定制了 NimbleGen SeqCap EZ Choice Library 靶向序列捕获产品作为本次 CAPP-Seq 的"筛选器",包含了 139 个相关基因的 521 个外显子和 13 个内含子,共约 125kb。 NimbleGen "筛选器"有效的把测序区段浓缩到整个基因组大小的 0.004%,使得后续超高深度测序得以实现。

CAPP-seq 是一个相当好的检测肿瘤基因突变的方法
参考文献
Aaron M Newman,et al. An ultrasensitive method for quantitating circulating tumor DNA With broad patient coverage. Nature Medicine.2014 May;20(5):548-54.